Extracto del libro "Lamarck y los mensajeros".
Autor: Máximo Sandín (Departamento de Biología. Universidad Autónoma de Madrid).
Los transportadores de información genética.
Los virus se consideran agentes patógenos que interfieren en el
funcionamiento normal de las células. Como ya se ha comentado,
su misterioso origen se solventa con la hipótesis de «un fragmen-
to de material genético escapado de algún genoma», que «de
alguna forma» adquirió una cubierta que hace posible que este
material genético traspase la membrana celular pudiendo inte-
grarse en el ADN celular, donde actúa como un gen ordinario. En
ocasiones permanece inactivo sin interferir en el funcionamiento
celular, pero otras veces se activa (frecuentemente por un estímu-
lo externo como pueden ser los rayos ultravioleta), y se divide
hasta causar la muerte de la célula huésped y la infección de otras
células. Los virus pueden subsistir fuera de la célula, pero en
estado inerte.
Un tipo especial de estos agentes infecciosos son los retrovirus.
Cuando penetra en la célula huésped su ARN es copiado en ADN
por una enzima propia del virus, la transcriptasa inversa, y esta
copia se integra en un cromosoma de la célula, en el que puede
permanecer en la forma que se conoce como provirus. El indivi-
duo infectado puede estar sano durante años, aunque porte en la
sangre los anticuerpos producidos contra el virus en el momento
de la infección.
Estas informaciones sobre los mecanismos de actuación de los
virus se obtienen fundamentalmente de estudios enfocados a su
aspecto patológico, aunque muchas de sus interesantes e inexpli-
cables propiedades pueden tener grandes repercusiones en la
comprensión de los mecanismos micro y macroevolutivos.
Experimentos de laboratorio han puesto de manifiesto que el
material genético, el ADN, tiene un comportamiento extremada-
mente estable. Se puede replicar más de cien millones de veces sin
equivocar un solo nucleótido de sus cadenas. Esta capacidad está
potenciada en la célula por los extraordinarios mecanismos de
reparación de errores mediante enzimas celulares específicos.
Sin embargo, los virus ARN varían (mutan) unas cien mil veces
más rápidamente que los virus ADN. El motivo fundamental es
que la enzima que controla la copia del ARN viral en ADN, la
transcriptasa inversa, «no sabe corregir» los errores de copia, con
lo que permite que, con frecuencia, se incorporen al ADN algunos
nucleótidos «inexactos». Si se tiene en cuenta la extremada esta-
bilidad del ADN, se puede pensar que ésta sería una importantí-
sima fuente de mutación.
Pero además existen otras fuentes de variabilidad en los virus:
los genomas virales pueden recombinarse entre sí. Estas recom-
binaciones conllevan pérdidas o ampliaciones de ciertas regio-
nes, con lo que pueden aparecer variaciones genéticas bruscas, que
afectan a amplias regiones del genoma viral.
Esta capacidad de variación genética les convierte en unos
extraordinarios «transportadores» de información genética, con
propiedades biológicas diferentes según las variantes: se ha obser-
vado que pueden cambiar de especificidad tisular (celular), e
incluso pueden cambiar o atenuar su especificidad a nivel de
especie. Si a estas cualidades añadimos su capacidad de «capturar»
genes, las consecuencias evolutivas pueden ser sorprendentes. En
efecto: un virus integrado puede abandonar el genoma huésped
llevando consigo un segmento cromosómico, que integrará en el
de la nueva célula que infecte. Esto se ha podido comprobar en
casos de cáncer transmitido mediante virus que habían capturado
genes cancerígenos.
Esta transmisión horizontal de información genética sería una
forma de homogeneizar los genomas entre distintos grupos ani-
males (Fig. 20). De hecho, existen evidencias de esa transferencia
de información, en forma de transposones, entre insectos y pla-
telmintos (Planaria), pertenecientes a Phyla distintos (40).
En el caso de las variantes del virus del SIDA (un retrovirus),
resultó sorprendente que de los dos tipos conocidos hasta ahora,
el HIV-1 y el HIV-2 están, cada uno, más próximos a los virus de
ciertos simios de lo que están entre sí: el 1 a uno del chimpancé, y
el 2 al de un macaco (Macaca macúlala) y al del «mono verde»
(Cercocebus atys). La explicación de este extraño fenómeno (41) ha
quedado aclarada mediante la comparación de virus procedentes
de individuos portadores sanos en África, y los virus simiescos
(SIV), que no provocan enfermedad en su huésped: al parecer, se
trata de virus que han permanecido en el hombre y en los simios
desde hace muchísimo tiempo (como existen en muchos otros
mamíferos), y han sido las actividades humanas las que muy pro-
bablemente han favorecido su expansión y activación (42).
Solamente las propiedades y los comportamientos de los virus
hacen pensar en ellos como candidatos a un importante papel en
la evolución. Pero hay otros datos provenientes de investigaciones
aplicadas sobre ingeniería genética, que pueden complementar
esta posibilidad. Se trata del sorprendente mundo bacteriano.
Figura 20.- Las propiedades biológicas de los virus que hacen posible
su integración en un genoma celular, y su posterior salida de éste con
parte de material genético incorporado a su propio genoma, podrían
tener unas repercusiones evolutivas sorprendentes. En caso de que
dichos fragmentos de ADN tuvieran la posibilidad de complementarse
con su nuevo genoma huésped en función de la localización en que se
insertase, podrían producirse intercambios de información genética
entre distintas especies, que, a largo plazo, podrían llegar a producir una
homogeneización de sus genomas.
Los elementos «accesorios»
Las bacterias (los primeros organismos vivos) tienen, en
muchas ocasiones, en su interior unos «elementos genéticos acce-
sorios» que son estudiados y conocidos fundamentalmente como
portadores de resistencia a antibióticos y como vectores (sistema
de transferencia de información) en ingeniería genética.
Estos elementos, conocidos como plásmidos, tienen (cómo
no), un origen misterioso y unas propiedades que se podrían
calificar de extremadamente «lamarckianas». Son moléculas cir-
culares de ADN en doble cadena que se multiplican indepen-
dientemente dentro de las células y que se transmiten cuando
éstas se dividen. Sus características son las de un virus sin la cáp-
sula proteica y, al igual que éstos, sólo tienen actividad dentro de
las células (Fig. 21).
su integración en un genoma celular, y su posterior salida de éste con
parte de material genético incorporado a su propio genoma, podrían
tener unas repercusiones evolutivas sorprendentes. En caso de que
dichos fragmentos de ADN tuvieran la posibilidad de complementarse
con su nuevo genoma huésped en función de la localización en que se
insertase, podrían producirse intercambios de información genética
entre distintas especies, que, a largo plazo, podrían llegar a producir una
homogeneización de sus genomas.
Los elementos «accesorios»
Las bacterias (los primeros organismos vivos) tienen, en
muchas ocasiones, en su interior unos «elementos genéticos acce-
sorios» que son estudiados y conocidos fundamentalmente como
portadores de resistencia a antibióticos y como vectores (sistema
de transferencia de información) en ingeniería genética.
Estos elementos, conocidos como plásmidos, tienen (cómo
no), un origen misterioso y unas propiedades que se podrían
calificar de extremadamente «lamarckianas». Son moléculas cir-
culares de ADN en doble cadena que se multiplican indepen-
dientemente dentro de las células y que se transmiten cuando
éstas se dividen. Sus características son las de un virus sin la cáp-
sula proteica y, al igual que éstos, sólo tienen actividad dentro de
las células (Fig. 21).
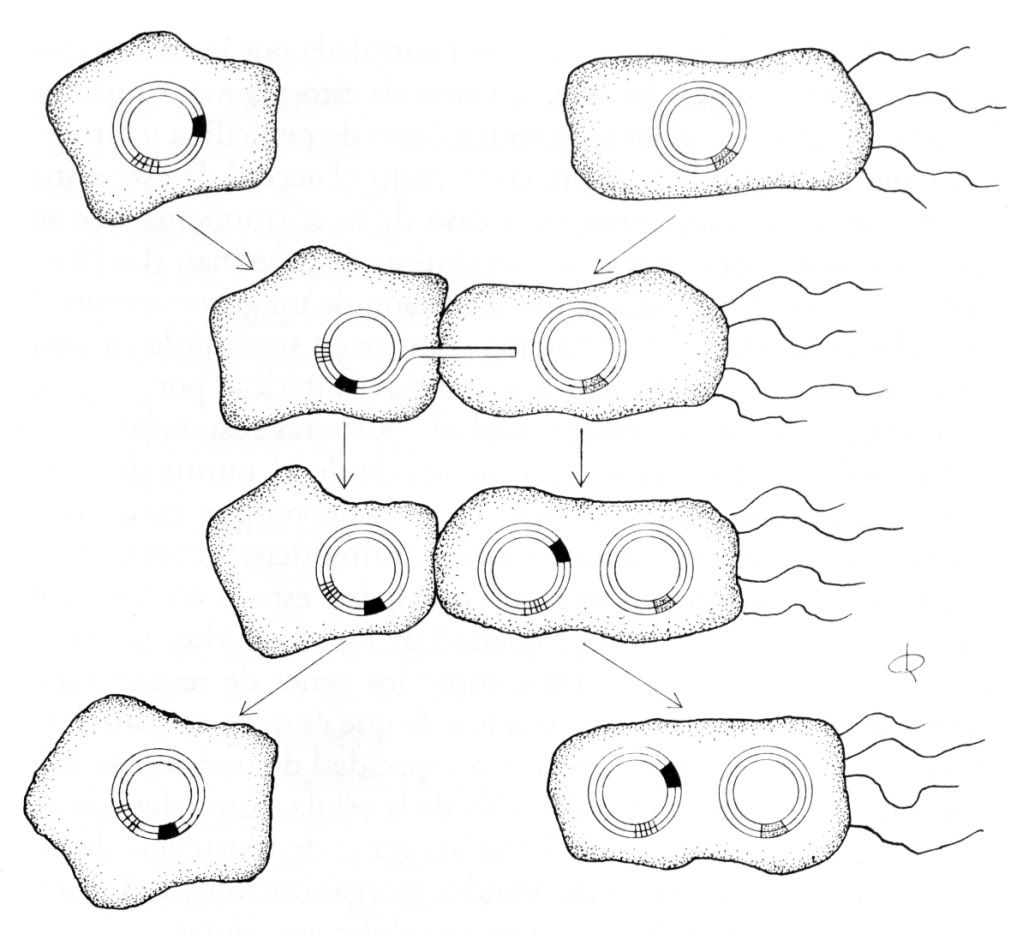
Figura 21.- La conjugación bacteriana, que inicialmente se inter-
pretó como «una especie de reproducción sexual» es un fenómeno no
sólo absolutamente inexplicable a la luz de las teorías neodarwinistas,
sino totalmente contradictorio con ellas. La transmisión de una infor-
mación genética, en muchos casos esencial para la supervivencia de la
receptora, se produce incluso entre géneros diferentes de bacterias, es
decir, se trata de una «cooperación» entre teóricas «competidoras». En
este caso se representa esquemáticamente la conjugación entre Escheri-
chia cali y Salmonella, que transfiere a la segunda un gen de resistencia a
la tetraciclina.
Estos extraños «organismos subcelulares» no pueden ser pro-
ducidos por las bacterias, sólo pueden ser adquiridos de una bac-
teria a otra, aunque no se sabe si siempre ha sido así, o cuándo se
adquirieron. El hecho es que, curiosamente, la información de los
plásmidos, que sólo constituye de un 1% a un 3% del genoma de
la célula, participa en la actividad bacteriana en funciones no
codificadas por el cromosoma: hacen posible la asimilación de
sustancias complejas, les confieren resistencia a tóxicos, entre
ellos los antibióticos, y son causantes de varias enfermedades en
animales y vegetales.
Pero además contienen la información para realizar una acti-
vidad muy especial: la conjugación bacteriana, que permite el
intercambio de plásmidos entre especies, e incluso géneros dife-
rentes de bacterias (43). Estos intercambios implican la transfe-
rencia de información genética de una especie a otra «competi-
dora» que, en muchos casos, hace posible a la receptora la propia
supervivencia. Es decir, estamos ante la existencia de unos carac-
teres adquiridos y transmisibles que son inexplicables como pro-
ducto de la Selección Natural.
Sus sorprendentes capacidades no se limitan a la cooperación
con la célula receptora. También le confieren nuevas propiedades.
La investigación sobre sus implicaciones en la adquisición por las
bacterias de resistencia a los antibióticos, ha permitido comprobar
la existencia de un mecanismo por el cual pueden ir acumulando
diferentes genes.
La resistencia a los antibióticos es controlada por los plásmidos
de tres maneras, según las características de éstos: la más simple es
la destrucción directa del antibiótico (caso de penicilina y estrep-
tomicina), la segunda consiste en impedir el acceso de éste a sus
puntos de actuación, como es el caso de la eritromicina que se
une a los ribosomas deteniendo su síntesis de proteínas, (los plás-
midos los protegen produciendo la adición de un grupo metilo al
ARN ribosómico). El tercer sistema consiste en sustituir la enzima
sensible a los antibióticos o a sustancias sintéticas por otra de
igual función pero que no sea inhibida por estas sustancias.
La explicación de estos fenómenos desde el punto de vista
evolutivo es francamente difícil, sobre todo porque existe una
resistencia natural simultánea a varios antibióticos y compuestos
sintéticos con los que no tendrían que haber estado en contacto
jamás. Pero lo es más otra propiedad de los plásmidos: la trans-
posición de genes. Los estudios sobre los genes de resistencia a
antibióticos llevaron a la observación de que éstos se encontraban
en unos elementos genéticos con la capacidad de transferirse físi-
camente de un lugar a otro del ADN de la célula. Estos elementos,
los transposones, además de llevar los genes de resistencia de los
plásmidos, eran causantes de grandes reorganizaciones del mate-
rial genético bacteriano, e incluso en células eucariotas.
La forma de adquirir nuevos genes y reorganizar a los antiguos
para conseguir una información genética que se adecúe a las
necesidades del huésped, se ha podido deducir mediante experi-
mentos de clonación en plásmidos. Cuando se digiere un plás-
mido con una enzima «restrictasa», su ADN se separa en frag-
mentos con extremos cohesivos. Posteriormente se tratan los
fragmentos con la enzima «ADN ligasa» y los fragmentos se unen
formando nuevos plásmidos reordenados al azar, con nuevas
propiedades. Estos nuevos plásmidos pueden perder o ganar
grandes segmentos de ADN sin disminuir su viabilidad, pero
existe una región muy pequeña en su ADN que resulta contener
todas las funciones que hacen viable al genoma. Si se afecta esta
zona el plásmido es destruido, es decir, existe una pequeña por-
ción de material genético, llamado «bloque replicador» (que se
ha calculado en no más de 2000 pares de bases) inamovible,
que es capaz de replicar plásmidos hasta 50 veces mayores que ella.
En definitiva, se puede decir que las características y las pro-
piedades de los virus y su «variedad» en forma de plásmidos, así
como su evidente relación con el funcionamiento del ADN de las
células eucariotas, les convierten en más que probables protago-
nistas con un papel principal en la evolución de la vida a partir de
las bacterias. Una posible prueba adicional de esta evolución por
agregación sucesiva de diferente información genética —y no
sólo por duplicaciones y diversificaciones— es la extremadamen-
te llamativa conservación de secuencias genéticas ancestrales, que
se pueden identificar y seguir desde las bacterias hasta organismos
superiores.
Es más, podría llegarse a la aventurada (e intuitiva) hipótesis de
que las peculiaridades de los virus ARN, con su susceptibilidad
para los errores de copia y sus efectos en las reordenaciones cro-
mosómicas, les hacen sospechosos de haber tenido una impor-
tante implicación en los procesos microevolutivos, mediante
pequeñas mutaciones y reordenaciones cromosómicas, y tam-
bién posibles causantes de especiaciones, es decir, «variaciones
sobre un mismo tema».
En cuanto a los grandes y bruscos cambios de organización
que, evidentemente, han tenido lugar en los procesos macroevo-
lutivos, los virus ADN, con su extremada estabilidad, pero con
igual capacidad de infección simultánea de organismos e integra-
ción en sus genomas, pueden justificar un papel menos cons-
tante, pero de mayor importancia.
Los virus endógenos
Para apoyar esta —posiblemente frágil— hipótesis, habría que
conceder a estas secuencias víricas el ya mencionado calificativo de
«subrutinas» de la vida. Pero esta categoría, quizás un tanto pre-
tenciosa, puede validarse a partir de datos obtenidos muy recien-
temente.
En la actualidad, se han identificado en el genoma de distintos
organismos unas proporciones variables de ADN denominadas
«virus endógenos». Existen diferentes grupos, y «la mayoría» se
consideran derivados de virus exógenos que infectaron las diversas
especies animales en el pasado, y han pasado a ser endógenos
mediante su inserción en células germinales. Algunos son verda-
deros «fósiles genéticos»; son provirus antiguos que han sufrido
múltiples mutaciones, aunque todavía es posible relacionarlos
con algunos retrovirus actuales. Hasta ahora se han identificado
claramente miles de copias de origen vírico, que constituyen más
del 0,5% del ADN total en aves y mamíferos (44). El problema está
en el ADN de origen viral que no se puede reconocer en la actua-
lidad por su divergencia con el tiempo o por proceder de virus
desconocidos. Pero si tenemos en cuenta la enorme posibilidad de
que éste sea el origen de, al menos, las secuencias repetitivas y los
elementos transponibles, la proporción sería considerable.
De hecho, a pesar de que la investigación no está orientada en
este sentido (identificar secuencias víricas implicadas en rasgos de
importancia evolutiva), los trabajos en torno a su carácter pato-
lógico están empezando a producir resultados reveladores. En un
estudio sobre las infecciones por retrovirus humanos (HTLV) en
Japón, se ha podido comprobar la existencia de asociación entre
individuos afectados por HTLV-1 y los antígenos de histocompa-
tibilidad (HLA) A26 y B62 (éste es un complejo polimorfismo de
aparición y mantenimiento inexplicable para la Teoría Sintética).
En una investigación encaminada a identificar los genes que
controlan el desarrollo embrionario del ratón (45), la inserción del
retrovirus de Moloney en un punto del genoma del óvulo provo-
có una mutación en el color del pelo, que desaparecía cuando el
virus abandonaba el genoma.
Si ampliamos observaciones de este tipo a los elementos retro-
transponibles, como son los UNE (long inserted element) de
mamíferos, encontramos pistas de mayor significación evolutiva:
el tipo L-l de éstos forma cerca del 5% del genoma humano, con
más de 50.000 secuencias, de las que unas 4000 son de gran
tamaño (sobre 6000 pares de bases). Se ha comprobado (46) que
una clase de éstos, la L-l.2, codifica una proteína con actividad de
transcriptasa inversa, que es necesaria para la movilidad de diver-
sos tipos de retroelementos en mamíferos. Estas actividades no
sólo están relacionadas con aspectos patológicos (hipercolestero-
lemia familiar, hemofilia, enfermedad de Tay-Sachs...), sino tam-
bién con ciertas funciones importantes de complicada adquisición
evolutiva, como la formación y el funcionamiento del cristalino
del ojo de mamíferos.
Otro logro evolutivo de tanta complejidad como trascendencia
en la aparición de los mamíferos, la placenta, tiene retroelementos
implicados en su funcionamiento. Hasta hace poco se pensaba
que el efecto ejercido por un gen no dependía de su procedencia
paterna o materna, pero se han encontrado pruebas de que
machos y hembras ejercen una impronta (imprinting) sobre los
genes que transmiten. En mamíferos placenrarios los genes pater-
no y materno contribuyen de forma diferente, pero complemen-
taria, al desarrollo embrionario. Sin la impronta de la madre el
embrión es anormal; sin la impronta del padre, la placenta no
llega a desarrollarse.
Este mecanismo parece estar implicado en el propio origen y
evolución de la placentación, por una parte, para que la propia
madre acepte el desarrollo de un cuerpo extraño dentro de ella, y
por otra, para limitar este desarrollo impidiendo la invasión de los
tejidos maternos (47). Según datos recientes, este fenómeno habría
sido inducido por la inserción de partículas retrovirales defectivas
del tipo I.A.P. (48, 49).
La búsqueda de nuevas actividades (en procesos como los
mencionados), en todos estos tipos de secuencias de ADN (cuya
condición ha de ser «no funcional» para que se cumplan las ecua-
ciones de la genética de poblaciones), puede ser fundamental
para la comprensión de los procesos evolutivos. Hasta el momen-
to, los descubrimientos esporádicos y derivados de enfoques «apli-
cados» no sólo les han reconocido actividades integradas en el
funcionamiento del genoma, sino que en algunos casos han reve-
lado características que les asignan una actividad de primer orden.
Por ejemplo, muy recientemente se ha podido comprobar la exis-
tencia de una transmisión genética de la memoria inmunitaria
(adquirida) de linfocitos. Los resultados de la investigación ponen
de manifiesto la existencia de un mecanismo por el cual retrovirus
endógenos pueden transportar genes de la linea somática a la
germinal (50).
Pero no sólo existen ya datos sobre una actuación que se
puede considerar indirecta, como de «transportadores» de infor-
mación, o responsables de alteraciones (mutaciones) genéticas. Se
acaba de publicar (5 de mayo de 1995) la primera prueba de que
una secuencia retroviral de la familia de retrovirus endógenos
RTVL-1 se expresa en distintas estirpes celulares humanas, entre
las que están los linfoblastos B, de enorme importancia inmuni-
taria (51).
También se ha comprobado (52) que andgenos de origen retro-
viral se expresan en las células trofoblásticas normales de la pla-
centa humana, con una misión muy significativa: forman parte
del proceso de diferenciación morfológica de dichas células.
Es decir, la actividad de los retrovirus endógenos no se limita a
inducir reorganizaciones o modificaciones de la expresión de los
genes «activos», (lo cual ya sería muy indicativo desde el punto de
vista evolutivo); también se expresan directamente como parte de
procesos biológicos fundamentales.
---------------------
40. GARCÍA, J.; BAYASCAS, J.R.; MARFANY, G.; MUÑOZ, A.M.; CASALI, A.;
BAGUNA, J.; SALO, E. «High copy number ofhighiy similar mariner-
like transposons in planarian (Platyhelminthe): Evidence for a trans-
phyla horizontal transfer». Molecular Biology and Evolution 12:3,
1995.p.421.
41. HUET, T. et al. «Genetic organization of a chimpanzee lentivirus
related to Hiv-1». Nature 545, 1990. p. 356.
42. RÜBSAMEN-WAIGMANN, H.; DIETRICH, U. «La genealogía de los
virus». Mundo Científico, vol. 11, no 117, 1991.
43. NOVICK, R.P. «Plásmidos». Investigación y Ciencia, no 53, 1981.
44. COFFIN, J. Sexta Conferencia Internacional sobre retrovirus humanos:
HTLV. Absecon, New Jersey, 1994.
45. JENKINS, A. «Dilute (d) coat colour mutation ofDBA/2J mice is asso-
ciated with the site of integration ofa ecotropic MuLV genome».
Nature 295, 1981.p.370.
46. MATHIAS, S.L.; SCOTT, A.F.; KAZAZIAN, H.H.; BOEKE, J.D.;
GABRIEL, A. «Reverse transcriptase encoded by a human transposa-
ble elements». Science254, 1991. pp. 1808-1810.
47. HALL, S.G. «Genetic imprinting: Review and relevance to human
diseases». Am. J. Hum. Genet. 46, 1990. pp. 857-873.
48. DUHL, D.M.S. et al. «Neomorphic agoutimutations in obese yellow
mice». Nature Genetics 8, 1994. pp. 59-65.
49. NEUMANN, B.; KUBICKA, P.; BARLOW, D.P. «Characteristics of
imprinted genes». Nature Genetics'), 1995. pp. 12-13.
50. ROTHENFLUH, M.S. «Hypothesis: A memory lymphocyte specific
soma-to-germiine genetic feedback loop». Inmunoglobulin and Cell
B¿ology73, 1995.pp.2, 174.
51. INDRACCOLO, S.; GUNZBURG, W.M.; LEIBMOSCH, C.; ERFLE, U.;
SALMONS, B. «Identification of three human sequences with vi-
ral superantigen-specific primers». Mammalian Genome, 6, 1995.
p.5.
52. LYDEN, T.W.; JOHNSON, P.M.; MWENDA, J.M.; ROTE, N.S. «Expres-
sion of endogenous HIV-I crossreactive antigens within normal
human extravillous trophoblast cells». Joumal of Reproductive Inmu-
nology,28, 1995. p. 3.